유도진화는 단백질 공학에서 사용되는 자연 선택의 과정을 모방하여서 연구자가 의도하는 방향으로 단백질이나 염기 서열을 변화시키는 기술입니다. 이것은 처음에 유전자에 의도적으로 돌연변이를 주어서, 그 중 원하는 기능을 가진 것들을 선택하여, 그 것의 수를 증폭시키는 과정을 거칩니다. 이 기술은 살아있는 세포에서 in vivo 로도, 혹은 in vitro 로도 쓰여질 수 있습니다. 유도진화는 단백질 공학 분야에서 단백질을 조작하는 방법으로도, 또한 실험실에서 진화 과정을 연구하는 방법으로도 사용됩니다.
유도진화의 역사
초기 단계의 유도진화는 Barry Hall 의 주도 하에 1970년도에 시작하여 단백질 공학 기술의 일환으로 개발되었습니다. 1990년도 부터는 이 기술로 2018년 노벨 화학상을 받은 Frances Arnold 가 이를 크게 개선하여 과학계에 알리는 데에 공헌하였습니다. 유기 용매에서 안정적인 효소를 개발한 선행 연구를 바탕으로, Chen 과 Arnold 는 무작위의 돌연변이를 일으킨 다음, 이 중 적합한 돌연변이를 가진 것들을 유기용매 안에서 효소 작용이 일어나는 것으로 선별하는 원리를 적용하였습니다. 이러한 유도진화 기술은 유전자 조작 및 선별 기술 발전과 함께 1990년 대에 크게 유행하였습니다. 유도진화 기술로 2018년에 Frances Arnold, George Smith 그리고 Gregory Winter 에게 노벨 화학상이 수여되었습니다.
유도진화의 원리
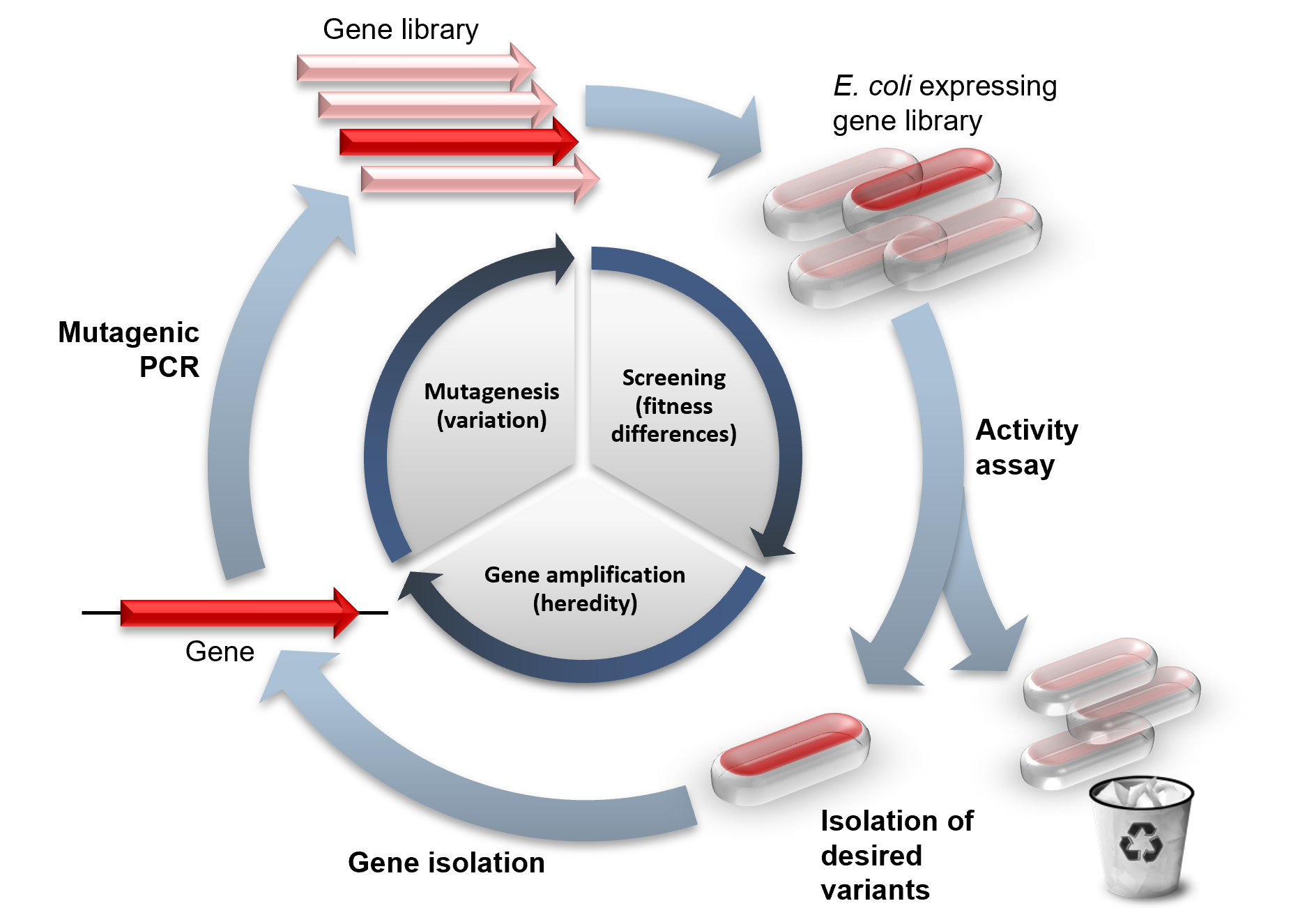
유도진화는 연구실 환경에서 자연 진화를 모방합니다. 진화에는 개체 간 적응도의 다양성 (fitness differences), 그 다양성에 기반한 선택 (selection), 다양성의 상속 (heritability) 등의 세가지 과정이 필요합니다. 유도진화에서는, 하나의 유전자가 돌연변이 생성(mutagenesis), 선별 과정(screening), 증폭(amplification)의 단계를 거쳐서 진화합니다. 이 단계들은 보통 매 단계마다 가장 적합한 변이 형질을 골라서 반복되어서 순차적으로 개선된 형질을 선별합니다.
유도진화 실험의 성공 여부는 전체 라이브러리 크기에 직접적으로 좌우됩니다. 이것은 더 많은 변이 형질을 생성하는 것이 원하는 형질을 찾게될 확률이 높기 때문입니다.
다양한 형질 생성
유도진화의 첫번째 단계는 다양한 유전자 라이브러리를 생성하는 것입니다. 무작위의 염기서열이 가진 경우의 수는 방대하지만 (100 아미노산으로 이루어진 단백질은 염기서열로 보면 10130 가지의 경우의 수가 있다), 이 중 기능하는 단백질의 수는 아주 적습니다. 자연선택에서도, 인위적인 진화로도 이렇게 많은 염기서열을 모두 테스트해볼 수는 없는 일입니다. 물론, 자연 진화는 이러한 변이 서열들이 이미 기능하는 단백질 염기서열과 비슷한 염기서열을 가집니다. 그리고 유도진화에서는 이미 기능을 가진 유전자에 돌연변이를 줌으로써 이를 모방합니다.
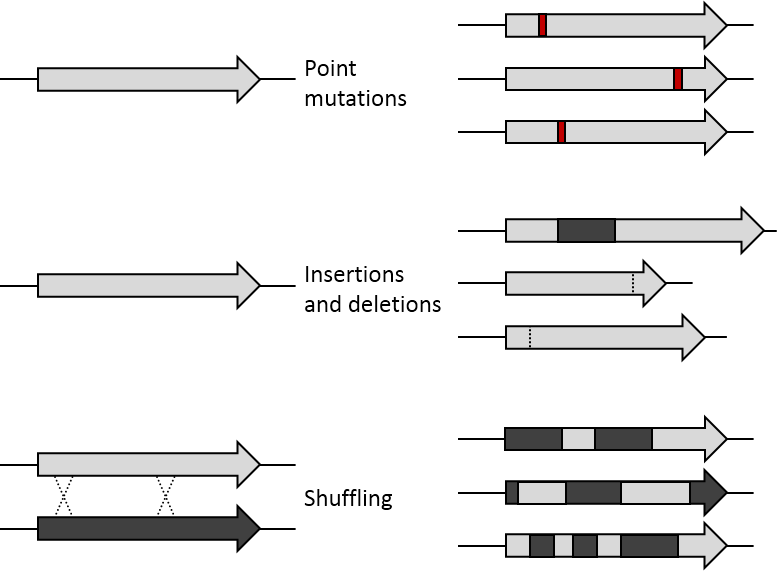
기존의 유전자는 화학 물질이나 error prone PCR 등의 방법으로 무작위의 점변이(random point mutation) 또는 transposon을 이용한 삽입(insertion) 및 삭제(deletion)으로 돌연변이가 만들어집니다. 유전자 재조합은 대략 70%이상 염기서열이 비슷한 여러 염기 조각들을 사용한 DNA shuffling으로 모방할 수 있습니다. 그로 인해 유전자의 특정한 위치에서 체계적으로 교환이 일어날 수 있습니다. 생성된 라이브러리는 이러한 변이 방법에 따라 기능을 하는 돌연변이의 비율이 달라집니다. 만약 특정한 유전자만이 돌연변이되어 발현되더라도 개체 안에서 나머지 유전자들은 항상성을 어느정도 유지할 수가 있습니다.
적응도 차이의 선별
대부분의 돌연변이는 개체에 해로우며 그로 인해 돌연변이가 생성된 라이브러리는 거의 대부분 감소된 적응도를 보입니다. 그러므로 목표로 하는 형질을 가진 이로운 돌연변이를 찾기위한 적응도의 대량 측정 방법은 아주 중요합니다. 이러한 변이 형질을 찾기 위해서는 대표적으로 두가지 유형이 있습니다. 선택(selection) 시스템은 단백질 기능을 유전자의 생존에 직접적으로 연결시키는 반면에, 검별(screening) 시스템은 각각의 돌연변이를 테스트하여 양적인 수치를 낸 후, 원하는 형질을 가진 돌연변이 집단을 정렬하여 임의의 기준 이상의 그룹으로 구분합니다. 선택과 검별 시스템은 둘다 살아있는 세포에 in vivo로 이루어질 수 있으며 세포 없이 단백질이나 RNA에 직접 in vitro로도 적용될 수 있습니다.
in vivo 시스템의 경우에, 각각의 세포(주로 박테리아 혹은 효모)는 서로 다른 여러가지의 변이 형질을 가진 플라스미드가 주입됩니다. 이러한 방법으로, 각 세포 간에는 다른 유전체는 동일하며, 변이 형질을 가진 유전자 만이 상이하게 됩니다. 세포들은 세포질 혹은세포 표면에서 목표 단백질을 발현하며 여기서 그 단백질의 기능을 테스트할 수 있습니다. 이 방식은 세포 환경 내에서의 현상을 지켜볼 수 있는 이점이 있습니다. 세포 없이 실험할 경우에 유도진화는 인공 발현 및 전사(in vitro transcription translation)을 이용하여 단백질 혹은 RNA를 시험관 안에서 생성합니다. 이 방법은 온도, 용매 등의 다양한 환경을 좀 더 정확하게 조절하여 선택 조건을 바꿀 수 있는 장점 뿐만 아니라, 세포에 독성을 지니는 단백질도 생성할 수 있다는 이점이 있습니다. 게다가 in vitro 실험은 DNA가 세포에 주입될 필요가 없기 때문에 in vivo 실험에 비해 훨씬 많은 라이브러리 (1015개 까지) 생성할 수 있습니다.
효소 결합의 선택(selection) 시스템은 이론적으로 간단합니다. 목표 물질은 단단한 구조물에 붙어서 고정되고, 그 위로 형질 변환된 단백질 라이브러리를 흘러내립니다. 결합이 약한 단백질은 씻겨져 내려가고 붙어있는 단백질 형질들은 분리되어서 유전자를 분석합니다. 활성화된 촉매를 분리하기 위해서 고정된 효소 억제제를 사용하기도 합니다. 그러나 이러한 방법은 하나의 촉매만 선택할 뿐만 아니라 최종의 진정한 기질 반응을 관찰하지 못하는 한계가 있기도 합니다. 만약 효소 활동이 세포의 생존에 직접적인 영향을 미친다면, 세포의 생존 여부로 선택할 수 있기도 합니다. 선택 시스템은 검별 시스템에 비해 비용이 저렴하고 노동력이 많이 듭니다. 하지만 설계하기가 어려운 편이고 활동 여부를 제외한 활동 정도에 관한 정보는 알 수가 없습니다.
선택 시스템 대신에 검별(screening) 시스템을 사용할 수도 있습니다. 각각의 변이 유전자는 하나씩 발현되고 그것을 측정할 수가 있습니다. 변이 형질은 그 뒤에 정렬되어서, 실험자가 어떠한 변이형질들을 다음 단계에 쓸 것인지 정할 수가 있습니다. 그러나 가장 최신의 분석 기기라 하더라도 선택 시스템에 비해서는 적은 양의 라이브러리 만을 테스트 할 수 있다는 한계가 있습니다. 반면에 각 변이 유전자에 관해서는 상세한 정보를 얻을 수 있다는 장점도 있습니다. 이 데이터들은 또한 라이브러리의 활동성 분포를 분석하는 데에도 쓰입니다.
상속성의 확보
일단 기능을 발휘하는 단백질을 분리하고 나면, 그 유전자도 함께 분리하여서 유전 형질과 발현 형질을 연결하는 것이 중요합니다. 이것은 mRNA display와 같이 단백질과 그 mRNA가 전사 맨 끝 부분에 puromycin으로 서로 연결되는 방법일 수도 있으며, 세포의 특정 부분이나 유체 방울 안에 단백질과 그 mRNA를 같이 가두는 방법이 될 수도 있습니다. 이렇게 분리된 유전자 염기 서열은 PCR로 증폭되거나 박테리아에 형질 전환하여 삽입되기도 합니다. 하나의 염기서열 혹은 여러 종류의 염기 서열들이 그 다음 단계의 변이 생성에 이용될 수 있으며, 이리하여 분화-선택-증폭 순으로 반복되는 사이클은 단백질이 연구하는 의도하는 방향으로 변이되게 합니다.
단백질 인공설계 기술 과의 비교
유도진화의 장점
단백질의 인공설계는 그 단백질의 구조 뿐만 아니라 효소 작용 매커니즘에 관한 상세한 정보가 필요합니다. 단백질의 기능을 변화시키기 위해서 특정한 변화를 특정 지점 돌연변이 형성을 이용하여 주게됩니다. 이 방법의 단점은 그 단백질에 관한 많은 정보가 있다고 하더라도, 주어진 특정한 변화를 예측하기가 힘들다는 것입니다. 반면에 유도진화는 이렇게 단백질의 매커니즘에 관한 이해 및 돌연변이 결과의 영향도 미리 예측할 필요가 없습니다.
유도진화의 한계점
유도진화는 무작위로 생성된 많은 수의 변이된 형질들을 전부 측정할 대량의 분석 기술이 필요한 점에 그 한계가 있습니다. 이것은 유도진화 기술을 사용하기도 전부터 대규모의 기술 개발이 필요한 것을 의미합니다. 그리고 이러한 분석 기술은 대부분의 경우에 특정한 유도진화 실험에만 적용될 수 있어서, 새로운 유도진화 실험에는 다시 새로운 분석 기술을 개발하여야 합니다.
두 기술의 융합
위의 한계와 장점을 취합하여서 단백질 인공설계와 유도 진화를 융합해서 쓸 수 있는 기술이 연구되고 있는 중입니다. 무작위의 돌연변이를 생성할 때에, 적응성을 높이는 돌연변이는 아주 드물기 때문에, 수많은 변이 형질들을 탐색해야 합니다. '집중된 라이브러리(focused library)'는 이러한 무작위한 범위를 적응성을 높이는 방향으로 집중 시킬 수 있도록 시도합니다. 이 집중된 라이브러리를 생성하는 데에는 단백질의 어느 부분에 돌연변이를 줄 것인 지에 관한 지식이 어느정도 필요합니다. 예를 들어서, 한 효소의 기작과 반응에 관한 연구에는 그 효소의 반응 지점(active site)에 관한 정보가 필요합니다.
유도진화의 이용
유도 진화는 단백질 공학에서 단백질 인공 설계를 대신하여 자주 사용되지만, 효소의 진화 과정에 관한 탐구를 할 때에도 사용됩니다.
단백질 공학에서
단백질 공학 기술의 하나로, 유도진화는 세 분야에서 가장 유용합니다:
1. 고온이나 다양한 용매에서의 생명공학적 용도로 쓰이는 단백질의 안정성 향상
2. 치료용 항체의 결합성 및 새로 만들어진 효소의 활동성 향상
3. 기존 효소의 기작 특정성 변화
진화학 연구에서
자연 진화 연구는 전통적으로 현존하는 생물과 그 유전자에 기반하여서 이루어져 왔습니다. 하지만 이러한 연구는 화석(특히 고대 생물의 DNA)의 부족과 고대 환경에 관한 완벽하지 못한 이해로 인하여 한계가 있습니다. 유도진화는 진화현상을 각각의 유전자로 나누어서 통제된 환경에서 실험하는 것을 가능하게 합니다.
유도진화는 자연선택의 강도, 돌연변이의 정도, 주변 환경 등을 조절할 수 있습니다. 더군다나 진화 과정에서의 모든 유전자의 기록 또한 가능합니다. 이것은 진화 과정을 상세하게 측정할 수 있도록 해줍니다.
이 글은 위키피디아의 유도진화 / Directed Evolution 을 번역 및 편집한 것입니다. 원문은 여기서 찾으실 수 있습니다.
'Biological Science > 흥미로운 연구 모음' 카테고리의 다른 글
| [번역]천재 과학자를 자살로 이끈 후성유전학의 발견 (0) | 2018.04.20 |
|---|---|
| 유전체 연구로 바퀴의 끈질긴 생명력에 관한 비밀이 밝혀지다. (0) | 2018.03.23 |
| [번역] 후성유전을 이용한 희귀병 치료 (0) | 2018.03.09 |
| [번역]천식 환자의 면역 세포에 보이는 후성유전적 DNA 변화 (0) | 2018.03.08 |
| [번역] 공기의 질소를 흡수하는 유전자 조작 농작물 (0) | 2018.02.24 |